So erstellen sie ein restdiagramm in ggplot2 (mit beispiel)
Mithilfe von Residuendiagrammen wird beurteilt, ob die Residuen eines Regressionsmodells normalverteilt sind und ob sie Heteroskedastizität aufweisen.
Um ein Residuendiagramm in ggplot2 zu erstellen, können Sie die folgende grundlegende Syntax verwenden:
library (ggplot2) ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
Das folgende Beispiel zeigt, wie diese Syntax in der Praxis verwendet wird.
Beispiel: Erstellen eines Restdiagramms in ggplot2
Für dieses Beispiel verwenden wir den in R integrierten mtcars- Datensatz:
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3,460 20.22 1 0 3 1
Zuerst passen wir ein Regressionsmodell an, wobei wir mpg als Antwortvariable und qsec als Prädiktorvariable verwenden:
#fit regression model
model <- lm(mpg ~ qsec, data=mtcars)
Als Nächstes verwenden wir die folgende Syntax, um ein Residuendiagramm in ggplot2 zu erstellen:
library (ggplot2) #create residual plot ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 )
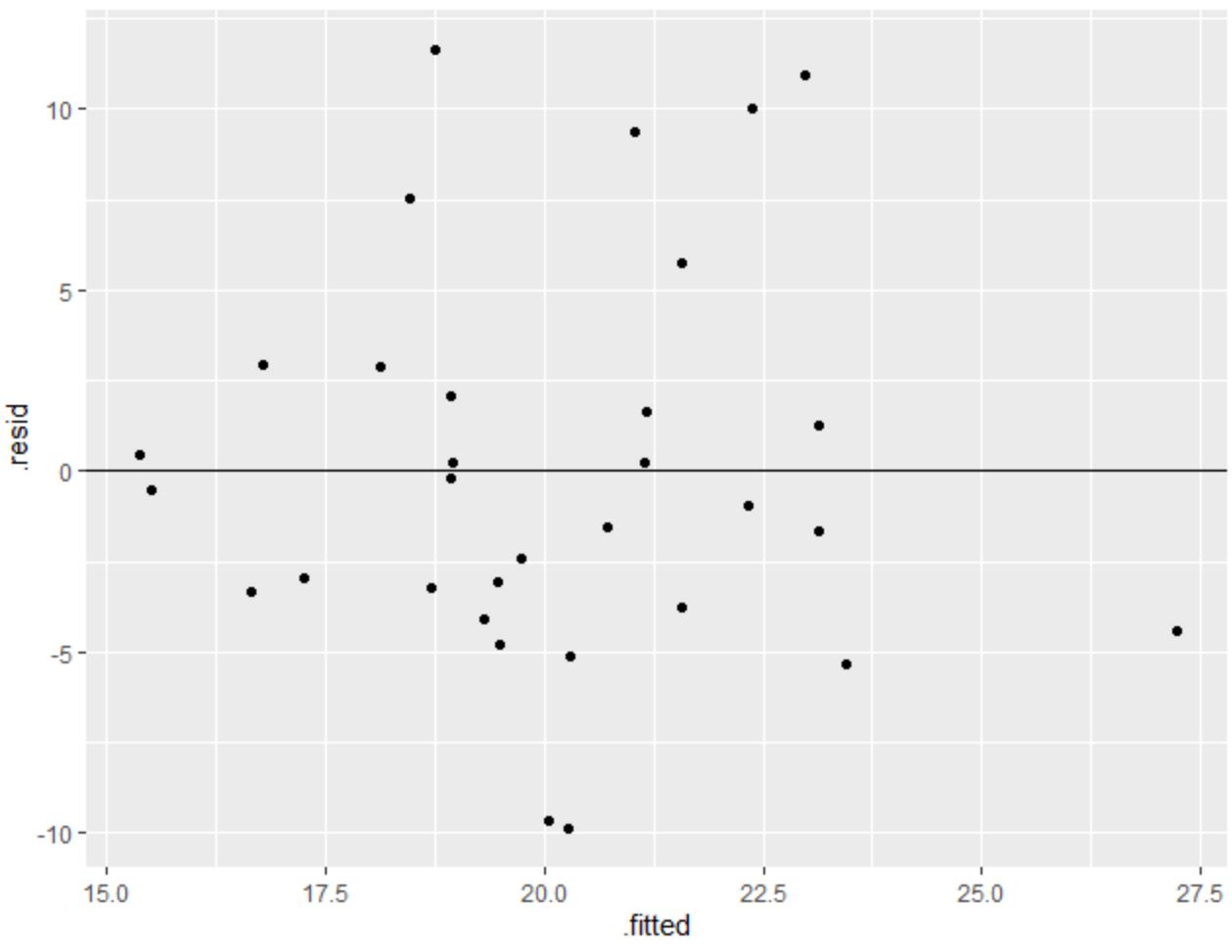
Die x-Achse zeigt die angepassten Werte und die y-Achse zeigt die Residuen an.
Die Residuen scheinen ohne klares Muster zufällig um Null herum verstreut zu sein, was darauf hindeutet, dass die Annahme der Homoskedastizität erfüllt ist.
Mit anderen Worten: Die Koeffizienten des Regressionsmodells sollten zuverlässig sein und wir müssen keine Transformation der Daten durchführen.
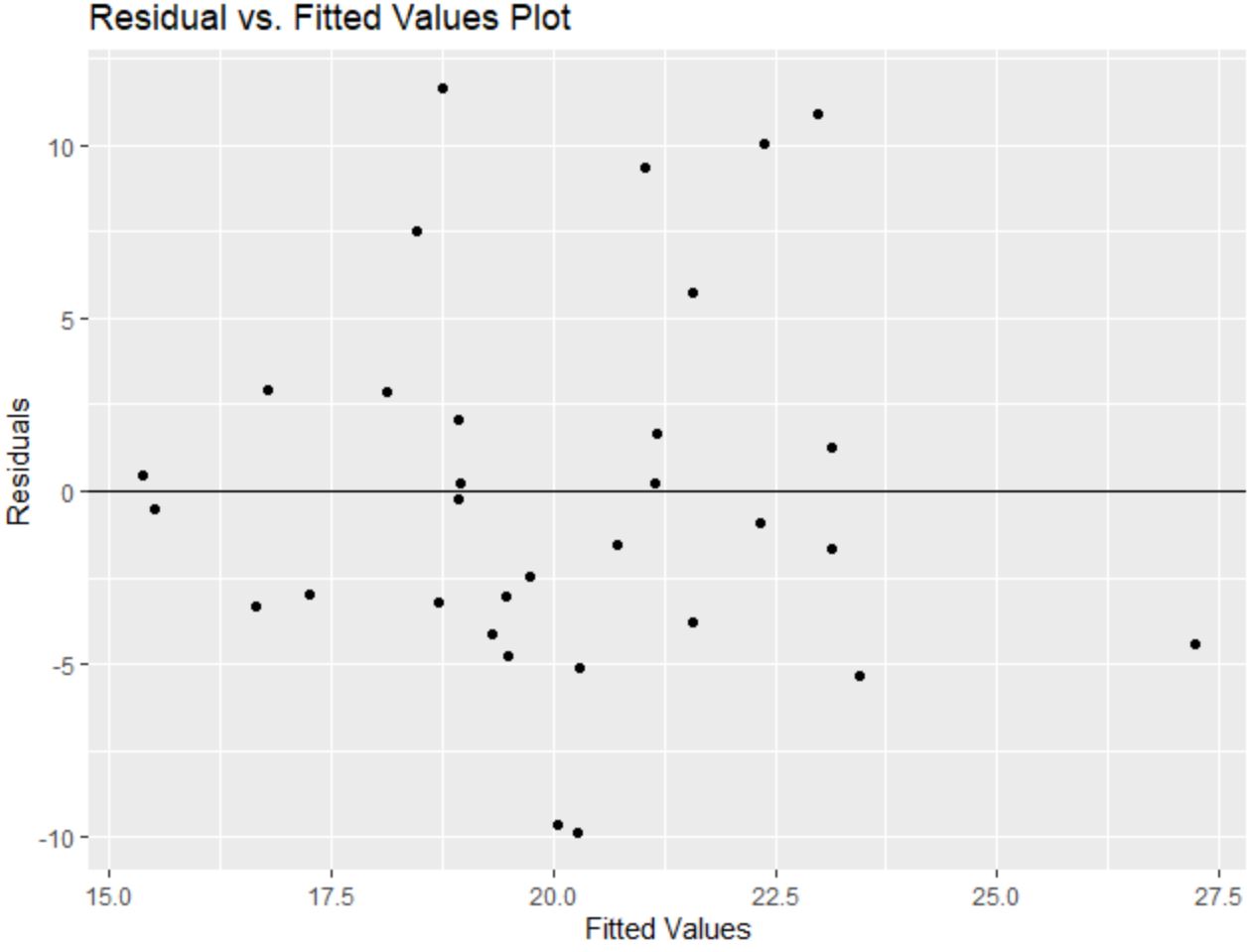
Beachten Sie auch, dass wir die Funktion labs() verwenden könnten, um dem Restplot einen Titel und Achsenbeschriftungen hinzuzufügen:
library (ggplot2) #create residual plot with title and axis labels ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0 ) + labs(title=' Residual vs. Fitted Values Plot ', x=' Fitted Values ', y=' Residuals ')
Zusätzliche Ressourcen
In den folgenden Tutorials wird erläutert, wie Sie andere häufige Aufgaben in R ausführen:
So berechnen Sie standardisierte Residuen in R
So berechnen Sie studentisierte Residuen in R
So erstellen Sie ein Histogramm von Residuen in R