Comment créer un tracé résiduel dans ggplot2 (avec exemple)
Les tracés des résidus sont utilisés pour évaluer si les résidus d’un modèle de régression sont normalement distribués et s’ils présentent ou non une hétéroscédasticité .
Pour créer un tracé résiduel dans ggplot2, vous pouvez utiliser la syntaxe de base suivante :
library(ggplot2) ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0)
L’exemple suivant montre comment utiliser cette syntaxe dans la pratique.
Exemple : création d’un tracé résiduel dans ggplot2
Pour cet exemple, nous utiliserons l’ensemble de données mtcars intégré dans R :
#view first six rows of mtcars dataset
head(mtcars)
mpg cyl disp hp drat wt qsec vs am gear carb
Mazda RX4 21.0 6 160 110 3.90 2.620 16.46 0 1 4 4
Mazda RX4 Wag 21.0 6 160 110 3.90 2.875 17.02 0 1 4 4
Datsun 710 22.8 4 108 93 3.85 2.320 18.61 1 1 4 1
Hornet 4 Drive 21.4 6 258 110 3.08 3.215 19.44 1 0 3 1
Hornet Sportabout 18.7 8 360 175 3.15 3.440 17.02 0 0 3 2
Valiant 18.1 6 225 105 2.76 3.460 20.22 1 0 3 1
Tout d’abord, nous allons ajuster un modèle de régression en utilisant mpg comme variable de réponse et qsec comme variable prédictive :
#fit regression model
model <- lm(mpg ~ qsec, data=mtcars)
Ensuite, nous utiliserons la syntaxe suivante pour créer un tracé résiduel dans ggplot2 :
library(ggplot2) #create residual plot ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0)
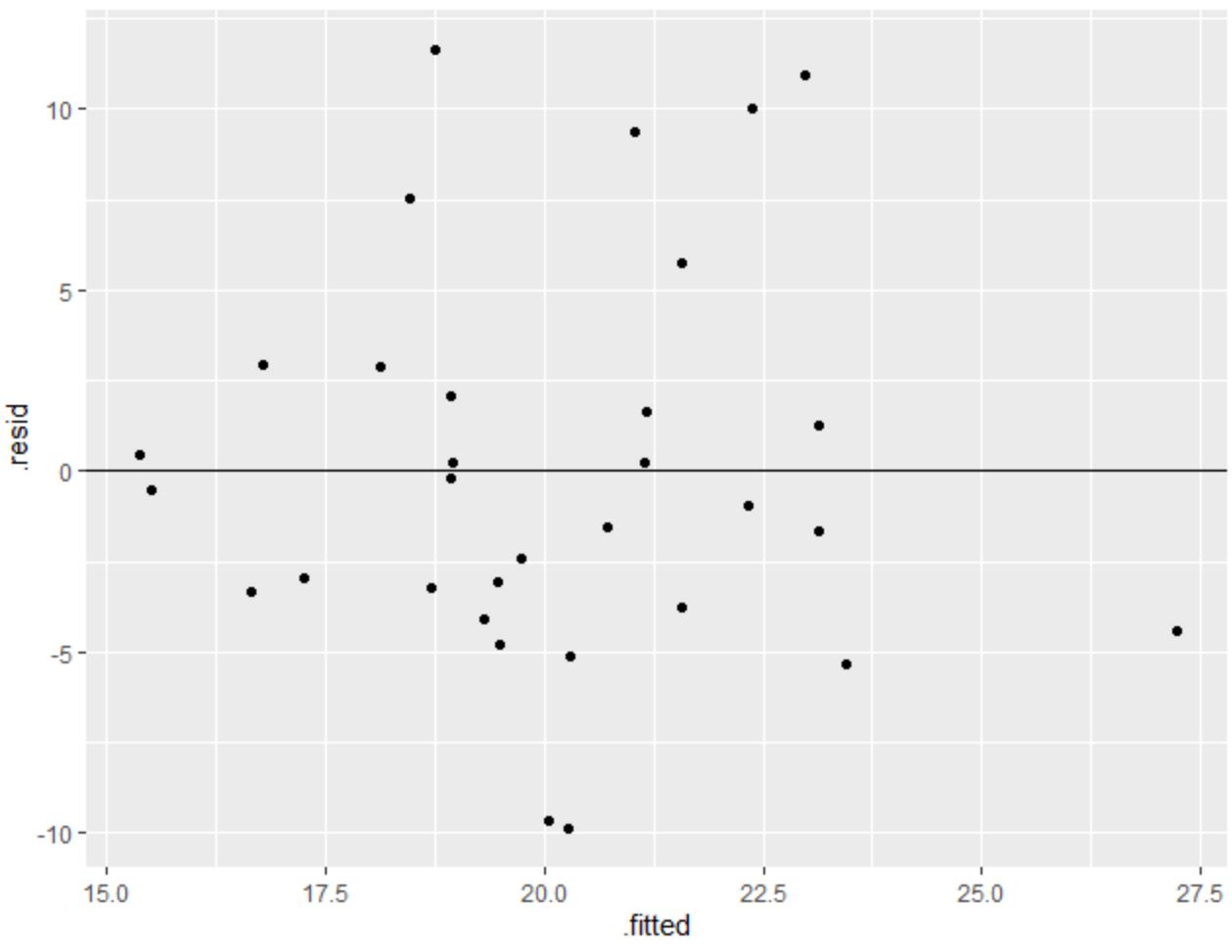
L’axe des x affiche les valeurs ajustées et l’axe des y affiche les résidus.
Les résidus semblent être dispersés de manière aléatoire autour de zéro sans motif clair, ce qui indique que l’hypothèse d’homoscédasticité est satisfaite.
En d’autres termes, les coefficients du modèle de régression doivent être fiables et nous n’avons pas besoin d’effectuer de transformation sur les données.
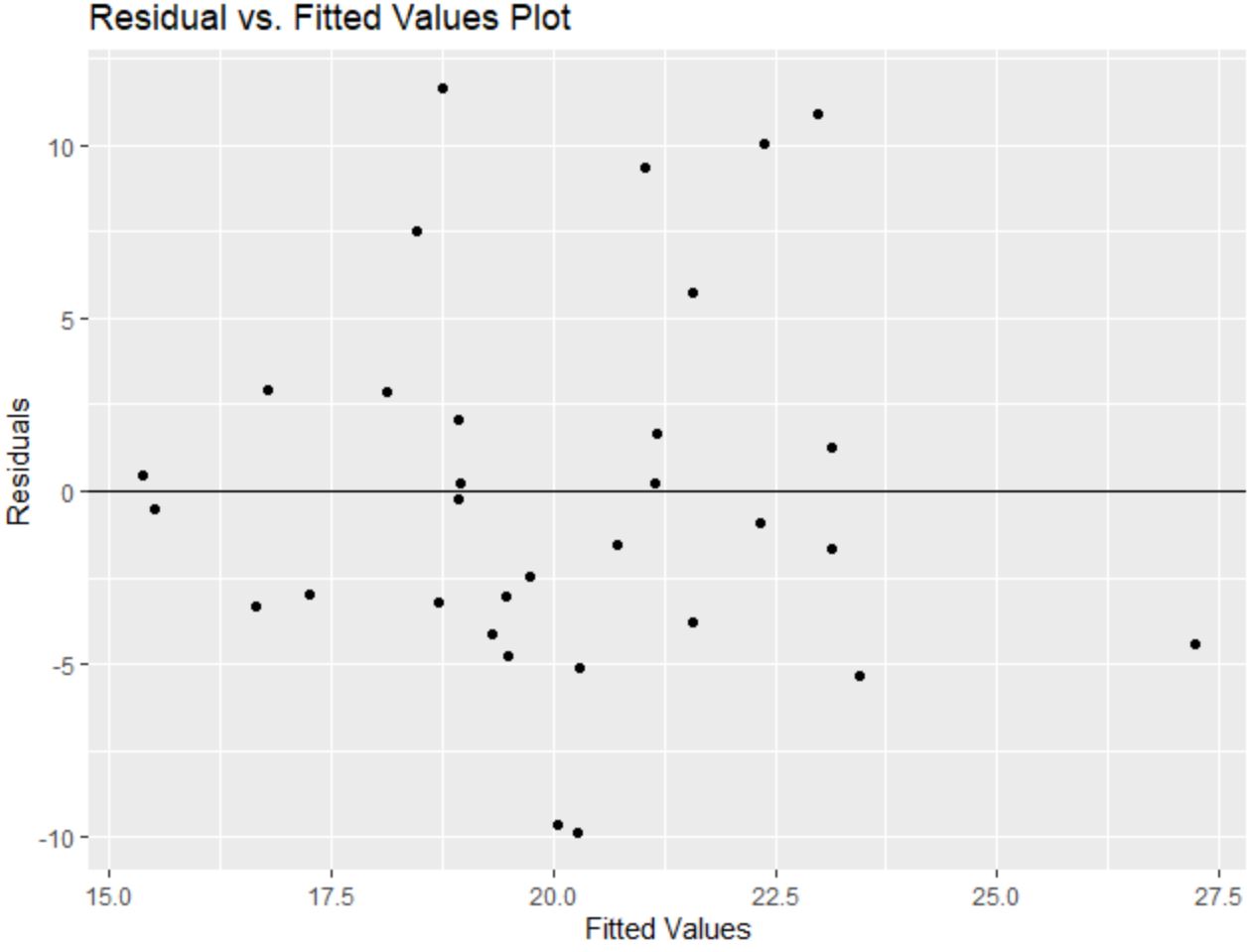
Notez également que nous pourrions utiliser la fonction labs() pour ajouter un titre et des étiquettes d’axe au tracé résiduel :
library(ggplot2) #create residual plot with title and axis labels ggplot(model, aes(x = .fitted, y = .resid)) + geom_point() + geom_hline(yintercept = 0) + labs(title='Residual vs. Fitted Values Plot', x='Fitted Values', y='Residuals')
Ressources additionnelles
Les didacticiels suivants expliquent comment effectuer d’autres tâches courantes dans R :
Comment calculer les résidus standardisés dans R
Comment calculer les résidus studentisés dans R
Comment créer un histogramme de résidus dans R
