Comment effectuer un test de Log Rank dans R
Un test de log-rank est le moyen le plus courant de comparer les courbes de survie entre deux groupes.
Ce test utilise les hypothèses suivantes :
H 0 : Il n’y a pas de différence de survie entre les deux groupes.
H A : Il y a une différence de survie entre les deux groupes.
Si la valeur p du test est inférieure à un certain niveau de signification (par exemple α = 0,05), nous pouvons alors rejeter l’hypothèse nulle et conclure qu’il existe suffisamment de preuves pour affirmer qu’il existe une différence de survie entre les deux groupes.
Pour effectuer un test de log-rank dans R, nous pouvons utiliser la fonction survdiff() du package survival , qui utilise la syntaxe suivante :
survdiff(Surv(heure, statut) ~ prédicteurs, données)
Cette fonction renvoie une statistique de test du chi carré et une valeur p correspondante.
L’exemple suivant montre comment utiliser cette fonction pour effectuer un test de log-rank dans R.
Exemple : test de classement de journal dans R
Pour cet exemple, nous utiliserons l’ensemble de données ovariennes du package de survie . Cet ensemble de données contient les informations suivantes sur 26 patients :
- Temps de survie (en mois) après avoir reçu un diagnostic de cancer de l’ovaire
- Si le temps de survie a été censuré ou non
- Type de traitement reçu (rx =1 ou rx = 2)
Le code suivant montre comment afficher les six premières lignes de cet ensemble de données :
library(survival) #view first six rows of dataset head(ovarian) futime fustat age resid.ds rx ecog.ps 1 59 1 72.3315 2 1 1 2 115 1 74.4932 2 1 1 3 156 1 66.4658 2 1 2 4 421 0 53.3644 2 2 1 5 431 1 50.3397 2 1 1 6 448 0 56.4301 1 1 2
Le code suivant montre comment effectuer un test de log-rank pour déterminer s’il existe une différence de survie entre les patients ayant reçu des traitements différents :
#perform log rank test
survdiff(Surv(futime, fustat) ~ rx, data=ovarian)
Call:
survdiff(formula = Surv(futime, fustat) ~ rx, data = ovarian)
N Observed Expected (O-E)^2/E (O-E)^2/V
rx=1 13 7 5.23 0.596 1.06
rx=2 13 5 6.77 0.461 1.06
Chisq= 1.1 on 1 degrees of freedom, p= 0.3
La statistique du test du Chi carré est de 1,1 avec 1 degré de liberté et la valeur p correspondante est de 0,3 . Puisque cette valeur p n’est pas inférieure à 0,05, nous ne parvenons pas à rejeter l’hypothèse nulle.
En d’autres termes, nous ne disposons pas de preuves suffisantes pour affirmer qu’il existe une différence statistiquement significative en termes de survie entre les deux traitements.
Nous pouvons également tracer les courbes de survie pour chaque groupe en utilisant la syntaxe suivante :
#plot survival curves for each treatment group plot(survfit(Surv(futime, fustat) ~ rx, data = ovarian), xlab = "Time", ylab = "Overall survival probability")
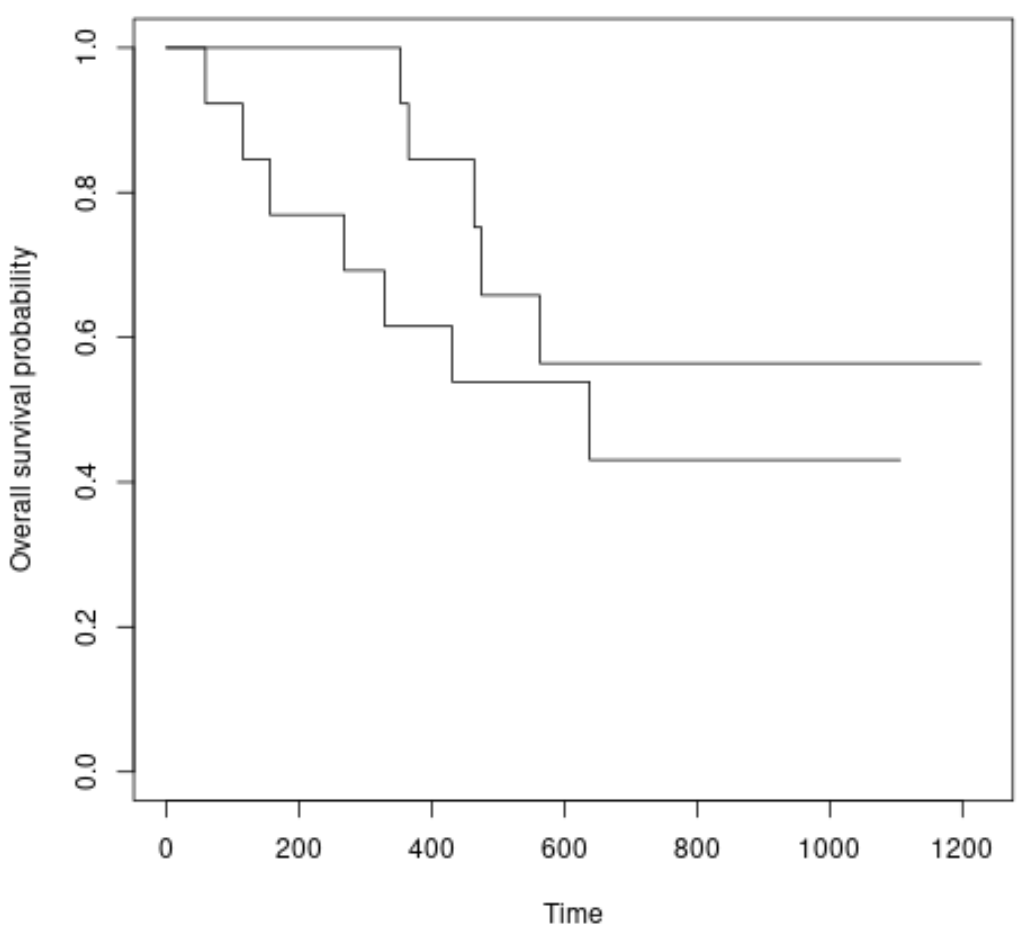
Nous pouvons voir que les courbes de survie sont légèrement différentes, mais le test du log-rank nous a indiqué que la différence n’est pas statistiquement significative.
