Comment effectuer une mise à l’échelle multidimensionnelle dans R (avec exemple)
En statistiques, la mise à l’échelle multidimensionnelle est un moyen de visualiser la similarité des observations dans un ensemble de données dans un espace cartésien abstrait (généralement un espace 2D).
Le moyen le plus simple d’effectuer une mise à l’échelle multidimensionnelle dans R consiste à utiliser la fonction cmdscale() intégrée, qui utilise la syntaxe de base suivante :
cmdscale(d, eig = FAUX, k = 2, …)
où:
- d : Une matrice de distance généralement calculée par la fonction dist() .
- eig : s’il faut ou non renvoyer les valeurs propres.
- k : Le nombre de dimensions dans lesquelles visualiser les données. La valeur par défaut est 2 .
L’exemple suivant montre comment utiliser cette fonction dans la pratique.
Exemple : mise à l’échelle multidimensionnelle dans R
Supposons que nous ayons le bloc de données suivant dans R qui contient des informations sur divers joueurs de basket-ball :
#create data frame df <- data.frame(points=c(4, 4, 6, 7, 8, 14, 16, 19, 25, 25, 28), assists=c(3, 2, 2, 5, 4, 8, 7, 6, 8, 10, 11), blocks=c(7, 3, 6, 7, 5, 8, 8, 4, 2, 2, 1), rebounds=c(4, 5, 5, 6, 5, 8, 10, 4, 3, 2, 2)) #add row names row.names(df) <- LETTERS[1:11] #view data frame df points assists blocks rebounds A 4 3 7 4 B 4 2 3 5 C 6 2 6 5 D 7 5 7 6 E 8 4 5 5 F 14 8 8 8 G 16 7 8 10 H 19 6 4 4 I 25 8 2 3 J 25 10 2 2 K 28 11 1 2
Nous pouvons utiliser le code suivant pour effectuer une mise à l’échelle multidimensionnelle avec la fonction cmdscale() et visualiser les résultats dans un espace 2D :
#calculate distance matrix
d <- dist(df)
#perform multidimensional scaling
fit <- cmdscale(d, eig=TRUE, k=2)
#extract (x, y) coordinates of multidimensional scaleing
x <- fit$points[,1]
y <- fit$points[,2]
#create scatter plot
plot(x, y, xlab="Coordinate 1", ylab="Coordinate 2",
main="Multidimensional Scaling Results", type="n")
#add row names of data frame as labels
text(x, y, labels=row.names(df))
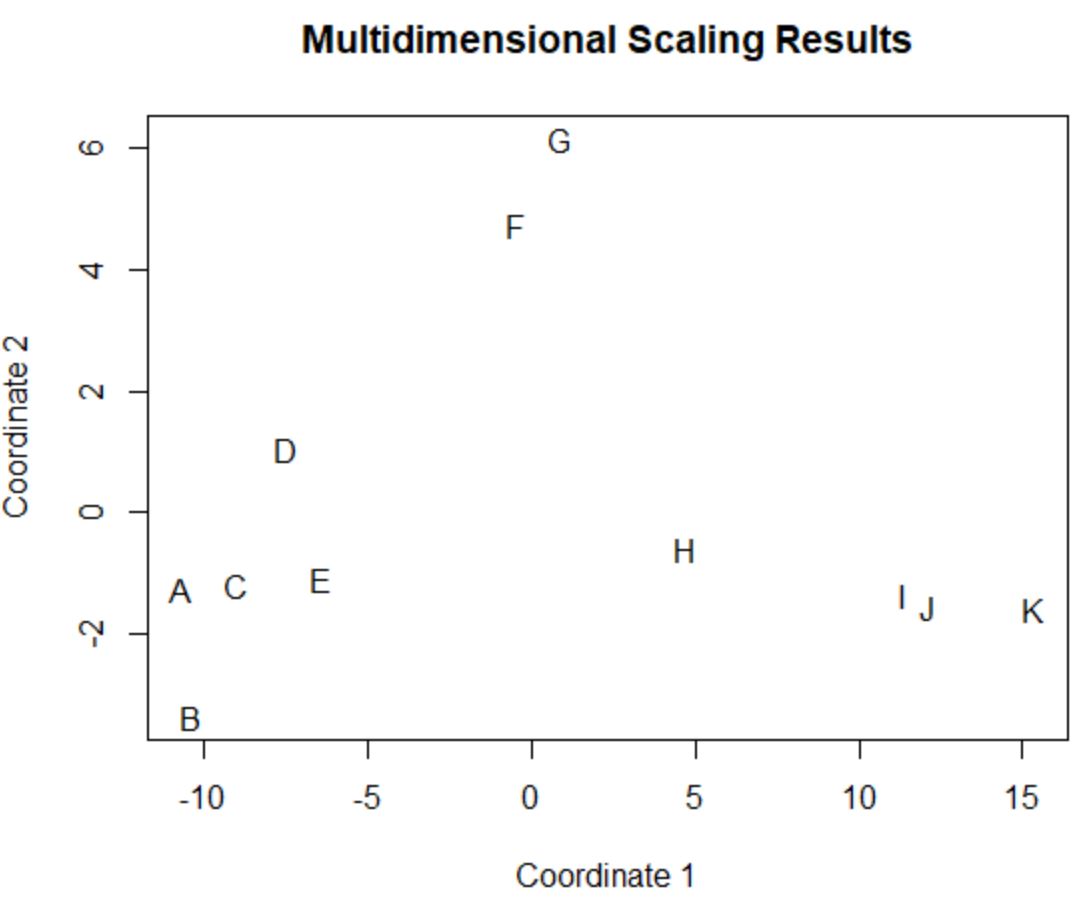
Les joueurs du bloc de données d’origine qui ont des valeurs similaires dans les quatre colonnes d’origine (points, passes décisives, blocages et rebonds) sont proches les uns des autres dans l’intrigue.
Par exemple, les joueurs A et C sont proches l’un de l’autre. Voici leurs valeurs à partir du bloc de données d’origine :
#view data frame values for players A and C df[rownames(df) %in% c('A', 'C'), ] points assists blocks rebounds A 4 3 7 4 C 6 2 6 5
Leurs valeurs pour les points, les passes décisives, les blocages et les rebonds sont toutes assez similaires, ce qui explique pourquoi ils sont si proches les uns des autres dans le tracé 2D.
En revanche, considérons les joueurs B et K qui sont éloignés l’un de l’autre dans l’intrigue.
Si nous nous référons à leurs valeurs dans les données originales, nous pouvons voir qu’elles sont assez différentes :
#view data frame values for players B and K df[rownames(df) %in% c('B', 'K'), ] points assists blocks rebounds B 4 2 3 5 K 28 11 1 2
Ainsi, le tracé 2D est un bon moyen de visualiser à quel point chaque joueur est similaire sur toutes les variables du bloc de données.
Les joueurs ayant des statistiques similaires sont regroupés à proximité tandis que les joueurs ayant des statistiques très différentes sont éloignés les uns des autres dans l’intrigue.
Notez que vous pouvez également extraire les coordonnées exactes (x, y) de chaque joueur de l’intrigue en tapant fit , qui est le nom de la variable dans laquelle nous avons stocké les résultats de la fonction cmdscale() :
#view (x, y) coordinates of points in the plot
fit
[,1] [,2]
A -10.6617577 -1.2511291
B -10.3858237 -3.3450473
C -9.0330408 -1.1968116
D -7.4905743 1.0578445
E -6.4021114 -1.0743669
F -0.4618426 4.7392534
G 0.8850934 6.1460850
H 4.7352436 -0.6004609
I 11.3793381 -1.3563398
J 12.0844168 -1.5494108
K 15.3510585 -1.5696166
Ressources additionnelles
Les didacticiels suivants expliquent comment effectuer d’autres tâches courantes dans R :
Comment normaliser les données dans R
Comment centrer les données dans R
Comment supprimer les valeurs aberrantes dans R
